Das VRC Projekt
Der Virtual Research Campus (VRC) ist eine moderne Datenmanagementplattform, die derzeit in Zusammenarbeit mit der Bioinformatik Würzburg (Prof. Thomas Dandekar), dem Servicezentrum Medizin-Informatik (SMI; Universitätsklinikum Würzburg) und dem Team der Core Unit FDM entwickelt wird. Inspiriert von erfolgreichen VRE-Modellen (e.g. VRE Charité, EBRAINS Projekt, CERN VRE) soll unser VRC Forschenden das Speichern, Austauschen und Auffinden von Daten, den sicheren Zugriff sowie eine innovative Nutzung und kollaborative Zusammenarbeit erleichtern.
Der VRC dient als umfassende Datenmanagementlösung, die Forschung und medizinische Projekte in Würzburg und assoziierten Forschungsgemeinschaften (standortübergreifend) unterstützt. Es ermöglicht die Speicherung, den Abruf, die Analyse und den Austausch von Daten sowie:
-
Metadaten-Management (automatische Erfassung und Zuordnung beschreibender Informationen)
-
Versionierung und Nachvollziehbarkeit (Tracking von Änderungen zur Sicherstellung der Reproduzierbarkeit)
-
Benutzer- und Zugriffsrechteverwaltung (flexible Rollen- und Gruppenmodelle zum Schutz sensibler Informationen)
-
Integration von Rechenressourcen und Analyse-Tools (Anbindung an HPC-Cluster, Container-Umgebungen und Webservices)
-
Workflows und Pipelines (Automatisierung komplexer Analyseprozesse über grafische oder skriptbasierte Abläufe)
-
Interoperabilität (Schnittstellen zu gängigen Datenbanken, Standards und API-basierten Diensten)
-
Suchfunktionen und Datenkataloge (Volltextsuche, Filtermechanismen, zentrale Datenübersicht)
-
Dokumentation und Leitfäden (eingebettete Wikis, Anleitungsvorlagen, Best-Practice-Beispiele)
-
Audit-Trails und Protokollierung (lückenlose Aufzeichnung aller Zugriffe und Aktivitäten)
Hierdurch werden Hindernisse für die Forschung beseitigt und Innovationen gefördert. Zum Beispiel soll der Datenaustausch zwischen Universitätskliniken und Universitäten erleichtert werden. Dafür ist geplant, ein GDPR-konformes System zu etablieren, um die Plattform auch für sensible klinische Daten nutzbar zu machen. Dabei müssen wir jedoch noch einige Hürden überwinden: Insbesondere geht es darum, technische Vorgaben (Verschlüsselung, Logging, Zugriffskontrolle) und organisatorische Prozesse (Auftragsdatenverarbeitung, Benutzerschulung, Risikobewertung) vollständig an die Anforderungen der Europäischen General Data Protection Regulation (GDPR) anzupassen. Unser Ziel ist es, Auftragsdatenverarbeitungsdienste für gesundheitsbezogene Forschungsprojekte unter vollständiger Einhaltung der GDPR bereitzustellen.
Die Zusammenarbeit mit Partnern wie dem NFDI-Netzwerk, der SMI, der Charité VRE, der Bioinformatik, dem Rechenzentrum und weiteren Universitäten und Kliniken stellt sicher, dass unsere Plattform den aktuellen Standards für Datenmanagement und Datensicherheit entspricht.
FAIR
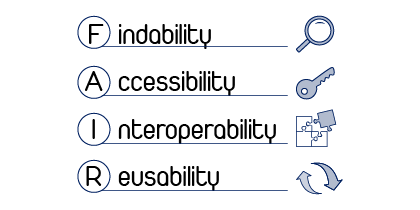
Der VRC ist fest in FAIR-Prinzipien verankert - Auffindbarkeit, Zugänglichkeit, Interoperabilität und Wiederverwendbarkeit. Er fördert die Interoperabilität mit internationalen Datengemeinschaften und bringt uns in den Einklang mit der Mission der Bavarian Health Cloud und der European Open Science Cloud (EOSC).
FAIR-Kernfunktionen des VRC:
- Auffindbar: Integrierte Erfassung, Indizierung und Speicherung von Metadaten mit Unterstützung für eindeutige Identifier und für flexible Datenorganisation.
- Zugänglich: Transparente Zugriffsrichtlinien und starke Authentifizierungs- und Autorisierungsverfahren.
- Interoperabel: Nahtlose Integration mit internationalen Datengemeinschaften und Unterstützung gemeinsamer Datenstandards.
- Wiederverwendbar: Annotierungs-Tools für eine umfassende Metadatenbeschreibung, interne Herkunftsverfolgung und Datalad-Support für die Herkunft von Code und Metadaten.
Architektur des Virtual Research Campus (VRC): Aus der Perspektive von Forschenden
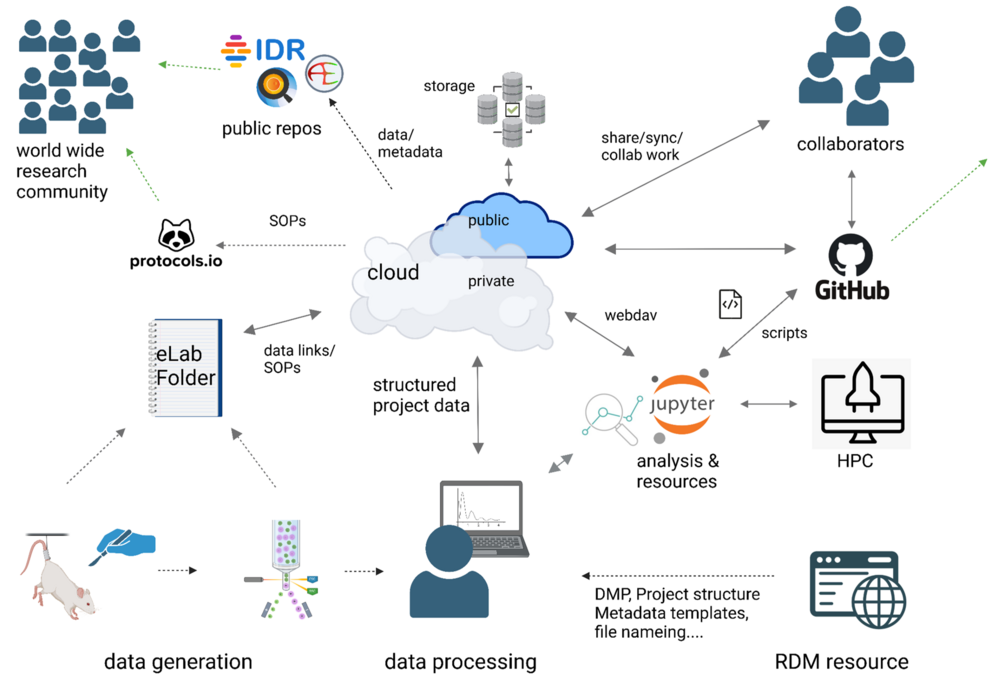
So einfach läuft Ihr Workflow im VRC der Uni Würzburg:
· Datengenerierung & Dokumentation
- Rohdaten entstehen im Labor (Tierversuch, Zellkultur, Messinstrumente).
- Dokumentation läuft über das elektronische Laborbuch (eLabFolder): SOPs, Protokolle, Parameter und Links zu jeder Datendatei sind zentral gespeichert.
- Metadaten & Skripte werden parallel erzeugt – so behalten Sie alle Informationen und Auswertungs-Scripts (Python, R, MATLAB…) beieinander.
· RDM-Knowledgebase & Standards
- Im zentralen RDM-Portal finden Sie Vorlagen für Ordnerstruktur, Dateinamen und Metadaten (nach NFDI-/EU-Richtlinien).
- FAIR-Checklisten zeigen, welche Formate und Versionierungstools (Git, Zenodo, Nextcloud) empfohlen sind.
· Organisiert lokal oder in der Cloud
- Legen Sie Ihre Projektordner nach Standard-Vorlage an (RawData, ProcessedData, Metadata, Analysis, Documentation).
- Lokal geht es genauso gut – Hauptsache: einheitlich & nach Vorgaben.
· Cloud-Sync & Datensicherung
- WebDAV-Anbindung spiegelt Ihre Ordner automatisch in die Private Cloud.
- Automatische Backups, Snapshots und versionskontrollähnliche Mechanismen schützen vor Datenverlust.
· GitHub für Skripte & Kollaboration
- Entwickeln Sie Ihre Auswertungs-Scripts direkt in einem GitHub-Repo.
- Versionierung, Pull Requests und Issue-Tracking erleichtern die Teamarbeit und Nachvollziehbarkeit.
· FAIR-Veröffentlichung
- Mit einem Klick exportieren Sie ausgewählte Datensätze und Metadaten in öffentliche Repositorien (z. B. IDR) – inklusive DOI, offenen Lizenzen und vollständiger Metadaten.
- Ihre Forschung wird weltweit FAIR-konform nachgenutzt.
Ihr Mehrwert
- Zeitersparnis: Reduzierung manueller Importe und Formatkonvertierungen
- Sicherheit: Rundumschutz für Ihre sensiblen Daten
- Reproduzierbarkeit: Jede Analyse ist jederzeit exakt rekonstruierbar
- Flexibilität: Nutzung Ihrer gewohnten Tools auf einer skalierbaren Plattform
- Transparenz: Metadaten und Protokolle bieten jederzeit vollen Einblick in Ihre Forschungs-Pipeline und erleichtern Suche und Integration mit anderen Daten.
Zukunftsarchitektur des VRC/VRE: Seamless Integration & State‐of‐the‐Art Orchestrierung
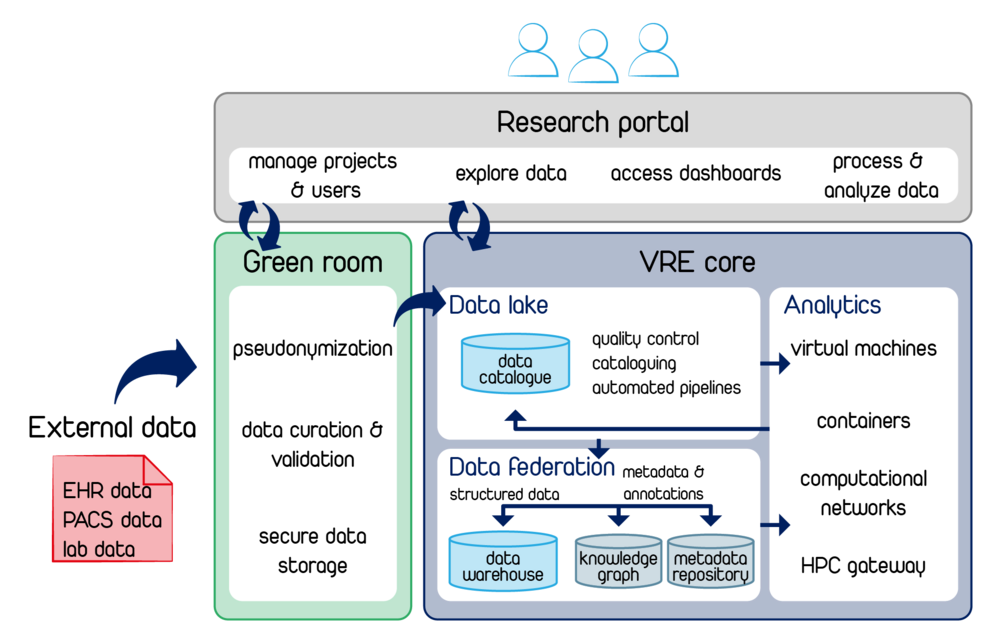
Die künftige Plattform setzt auf eine vollständig containerisierte Kubernetes-Umgebung, in der alle Dienste nahtlos und effizient zusammenspielen. Dadurch entsteht eine skalierbare, ausfallsichere Forschungsinfrastruktur, die den gesamten Datenlebenszyklus abdeckt.
- Modulares Webportal
- Warum? Einheitliche Zugriffskontrolle und Single-Sign-On für Forschende.
- Wie? Als eigener Kubernetes-Pod mit OAuth/SAML-Authentifizierung; kommuniziert über Service-Mesh verschlüsselt mit anderen Microservices.
- Green Room für sensible Daten
- Warum? Frühe Pseudonymisierung und Validierung sichern GDPR-/DSGVO-Konformität.
- Wie? In einem abgetrennten Kubernetes-Namespace laufen Verschlüsselungs- und Prüftools, die Rohdaten in ein geschütztes Storage-Backend schreiben.
- Automatisierte Datenaufnahme
- Warum? Heterogene klinische und Forschungsdaten müssen konsistent zusammengeführt werden.
- Wie? Mehrstufige Ingestion-Pipelines (Connector → Harmonizer → Ingestion-Service) verschieben Daten aus EHR/PACS/Laborsystemen in projektspezifische Data Warehouses und befüllen Metadaten-Kataloge automatisch.
- Projektspezifische Data Warehouses & Knowledge Graphs
- Warum? Isolation von Forschungsfällen und semantische Abfragen über Entitäten.
- Wie? Jeder Fall erhält ein eigenes Data Warehouse (z. B. PostgreSQL) und einen Knowledge-Graph-Container (z. B. Neo4j). Metadaten-Services aktualisieren Ontologien und Annotationen in Echtzeit.
- Containerisiertes Analyse-Framework
- Warum? Reproduzierbare, skalierbare Workflows für Deep Learning, Simulationen und Standardanalysen.
- Wie? On-Demand-Pods (JupyterLab, Nextflow) mounten Data Warehouses per CSI-Volume. Das HPC-Gateway leitet rechenintensive Jobs automatisch an Cluster weiter.
- FAIR & Metadaten-Management
- Warum? Auffindbarkeit, Interoperabilität und Wiederverwendbarkeit müssen durchgängig gewährleistet sein.
- Wie? Persistente Identifier und Metadaten halten Kataloge aktuell und ermöglichen Interoperabilität und Wiederverwendbarkeit. Provenance-Logs und Versionierung dokumentieren jeden Schritt.
- Kubernetes-Orchestrierung
- Warum? Automatisches Self-Healing, Rolling Updates und horizontale Skalierung sichern Verfügbarkeit und Leistung.
- Wie? Helm-Charts definieren Konfigurationen; Service-Mesh gewährleistet sicheren Datenverkehr; Event-Streaming koppelt Microservices locker.
- Reibungslose Kollaboration & Publikation
- Warum? Forschungsergebnisse sollen effizient geteilt und global sichtbar gemacht werden.
- Wie? Echtzeit Kollaboration via Webinterface/WebDAV/CSI-Volumes: Teammitglieder greifen gleichzeitig auf dieselben Daten und Dokumente zu. Automatische Veröffentlichung von freigegebenen Datensätzen in Repositorien (Zenodo, IDR) inklusive DOI- und Lizenz-Management.
Zusammenfassung:
In dieser Architektur sind alle Komponenten – von der Datenerfassung im Green Room über strukturierte Warehouses und containerisierte Analyse-Workspaces bis hin zur automatisierten Publikation – logisch und effizient miteinander verbunden. Kubernetes übernimmt die Orchestrierung, sodass die gesamte Plattform skalierbar, sicher und FAIR-konform betrieben werden kann.
Warum diese Architektur entscheidend ist
- Logische und effiziente Orchestrierung: Durch Kubernetes‐Orchestrierung und Service Mesh agieren alle Komponenten als einzelne Microservices, die bei Bedarf selbstständig starten, kommunizieren und ausfallen dürfen, ohne das Gesamtsystem zu beeinträchtigen.
- Nahtlose Daten‐ und Metadaten‐Flüsse: Automatisierte Pipelines verknüpfen Datenquellen, Curations‐Services und Analyse‐Workspaces durchgehend; Redundanzen und manuelle Importe entfallen.
- Hohe Skalierbarkeit und Ausfallsicherheit: Container‐basiertes Self‐Healing, horizontale Skalierung und Canary‐Deployments garantieren, dass bei steigender Nutzerzahl oder Ressourcennot das System dynamisch nachschaltet.
- Reproduzierbarkeit & FAIR‐Konformität: Versionierte Daten, lückenlose Provenance und persistent identifier sichern, dass Forschungsergebnisse dauerhaft nachvollziehbar und wiederverwendbar sind.